文章摘要
【关 键 词】 蛋白质生成、AI医疗、癌症治疗、模型架构、开源技术
微软开源的最新蛋白质生成模型BioEmu-1通过技术创新显著提升了动态蛋白质结构预测效率。该模型在单个GPU上每小时可生成数千种蛋白质结构,生成效率比传统分子动力学(MD)模拟提高数个数量级,为癌症等重大疾病治疗药物的研发开辟了新路径。其核心价值体现在快速解析复杂蛋白质动态变化,例如肿瘤抑制蛋白p53的突变体结构生成,可帮助医学研究者精准定位药物结合位点。
BioEmu-1的技术突破源于其独特的模型架构设计。基于改进的Distributional Graphormer框架,模型采用融合AlphaFold 2 evoformer模块的序列编码器,通过多层Transformer网络捕捉氨基酸序列的全局依赖关系。创新性的去噪扩散模型结合二阶积分方法,将去噪迭代次数大幅缩减,同时保持结构预测精度。训练过程中使用的2亿条蛋白质序列数据经过多级过滤,最终形成覆盖广泛蛋白质家族的5万个序列簇,确保了模型的泛化能力。
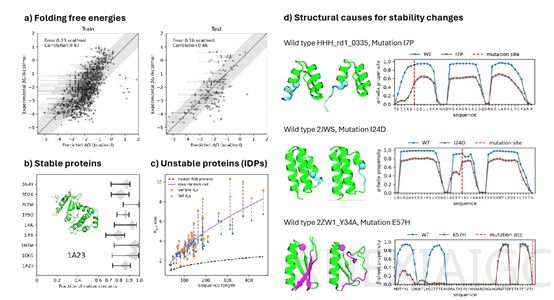
在应用验证方面,BioEmu-1展现出显著优势。针对快速折叠蛋白的测试显示,其完成折叠过程仅需数百GPU小时,而传统方法需数十万小时。模型生成结构与实验观测结果的平均绝对误差控制在0.15Å以内,对CATH域二级结构的预测准确率达到98%。在个性化医疗场景中,该模型可根据患者特定基因序列预测蛋白质结构变异,为定制癌症治疗方案提供支持,例如成功解析ACE2蛋白在不同构象下的动态特性。
实验数据表明,BioEmu-1对复杂蛋白结构的预测能力已接近实验观测水平。在处理Complexin II蛋白时,其预测的螺旋含量和回转半径与全原子力场计算结果误差小于5%。这些技术突破不仅加速了靶向药物开发进程,更标志着AI在生物大分子模拟领域实现了从结构预测向动态行为解析的跨越。随着模型开源,这一技术有望推动全球生物医药研究的协同创新。
原文和模型
【原文链接】 阅读原文 [ 1302字 | 6分钟 ]
【原文作者】 AIGC开放社区
【摘要模型】 deepseek-r1
【摘要评分】 ★★☆☆☆